
T淋巴細胞(T cell)和B淋巴細胞(B cell)主要負責適應性免疫應答,其抗原識別主要依賴于T細胞受體(T cell receptor, TCR)和B細胞受體(B cell receptor, BCR),這兩類細胞表面分子的共同特點是其多樣性,可以識別多種多樣的抗原分子。BCR的重鏈和TCR β鏈由V、D、J、C四個基因片段組成,BCR IGL鏈和TCR α鏈由V、J、C三個基因片段組成,這些基因片段在遺傳過程中發生重組、重排,組合成不同的形式,保證了受體多樣性。
傳統免疫組庫研究方式局限性較大,10x Genomics新推出的“單細胞免疫譜分析”利用其微流控芯片制備單細胞體系,選擇5’ 端接頭的通用引物和免疫分子恒定區的巢式引物進行V(D)J富集,實現在單細胞水平,對成對的重鏈和輕鏈(B細胞)或α和β鏈(T細胞)進行全長測序,為免疫組庫全面、系統的研究提供了高效的技術平臺,對疾病發生、發展的分子機制研究有重要的意義。

圖1. 10x Genomics單細胞系統
1 技術原理
10x Genomics單細胞免疫組庫測序是建立在GemCode技術上的微流體平臺,將帶有條形碼和引物的凝膠珠與單個細胞包裹在油滴中;接下來在每個油滴內,凝膠珠溶解,細胞裂解釋放mRNA,通過逆轉錄產生用于測序帶條形碼的cDNA。液體油層破壞后,cDNA一分為二,后續同時進行基因表達和免疫組庫文庫構建;其中TCR或者BCR的V(D)J序列通過設計在TCR或者BCR的C區域的巢式PCR引物進行富集。然后使用Illumina測序平臺對文庫進行測序檢測,即可一次性獲得大量單細胞的基因表達和免疫組庫數據,實現在單細胞水平同時對基因表達和免疫組庫的研究。
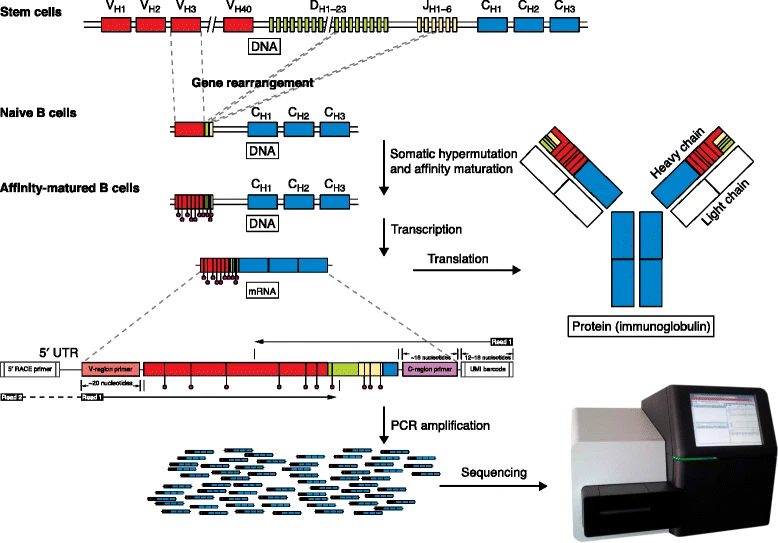
圖2. BCR測序技術流程[1]
2 樣本要求
◆ 樣品類型:新鮮人或小鼠細胞懸液、血液(全血、PBMC等);
◆ 細胞來源:血液提取、磁珠富集、流式分選、組織解離;
◆ 細胞大小:小于40μm;
◆ 細胞總量:細胞懸液,一個樣本細胞起始量約1x105個;
◆ 質量要求:細胞活性大于85%,理想濃度為1000個/μl(500~2000個/μl);
◆ 細胞培養基及緩沖液不能含有Ca2+和Mg2+等影響酶活性的物質。
3 實驗流程
利用10x Genomics平臺完成單細胞的分離、反轉錄成cDNA后,選擇不同的研究目的進行文庫構建:5’ 基因表達文庫、TCR的V(D)J文庫或和BCR的V(D)J文庫。將cDNA分成2份或3份同時進行TCR/BCR的V(D)J富集、文庫構建、測序和5’ 單細胞轉錄組文庫構建、測序,獲得每個細胞表達譜和V(D)J全長序列,獲得數據進行細胞亞群聚類、細胞表達特征和標記分子篩選等分析。

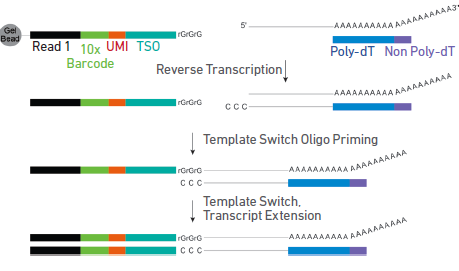

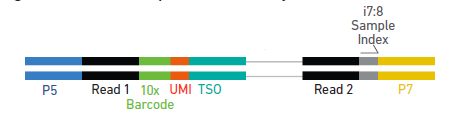
圖3. 實驗流程
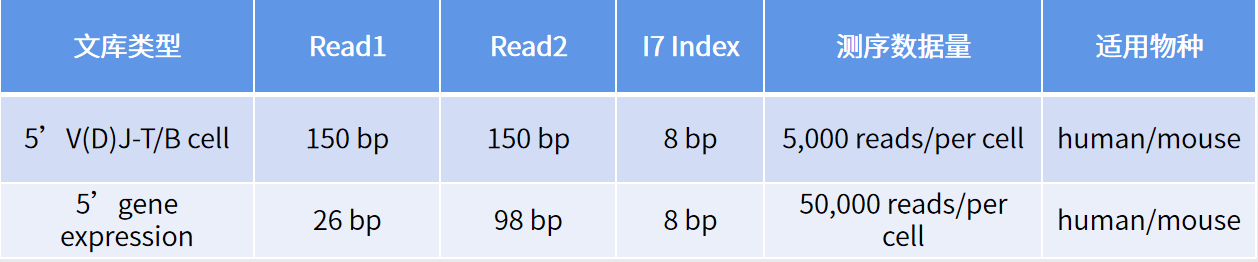
表1. 5’ 單細胞表達譜和V(D)J免疫組文庫及測序參數
4 分析內容
測序數據質量控制;
比對與注釋:樣本基本信息結果統計,樣本contig注釋信息結果統計,樣本Consensus序列注釋信息結果統計;
Clonotype分型;
特征分析:CDR3特征分析,V/J基因特征分析,V-J Paired特征分析;
多樣本分析:樣本間Clonotype比較,Overlapping Clonotype聚類分析,Overlapping Clonotype差異分析。
5結果展示
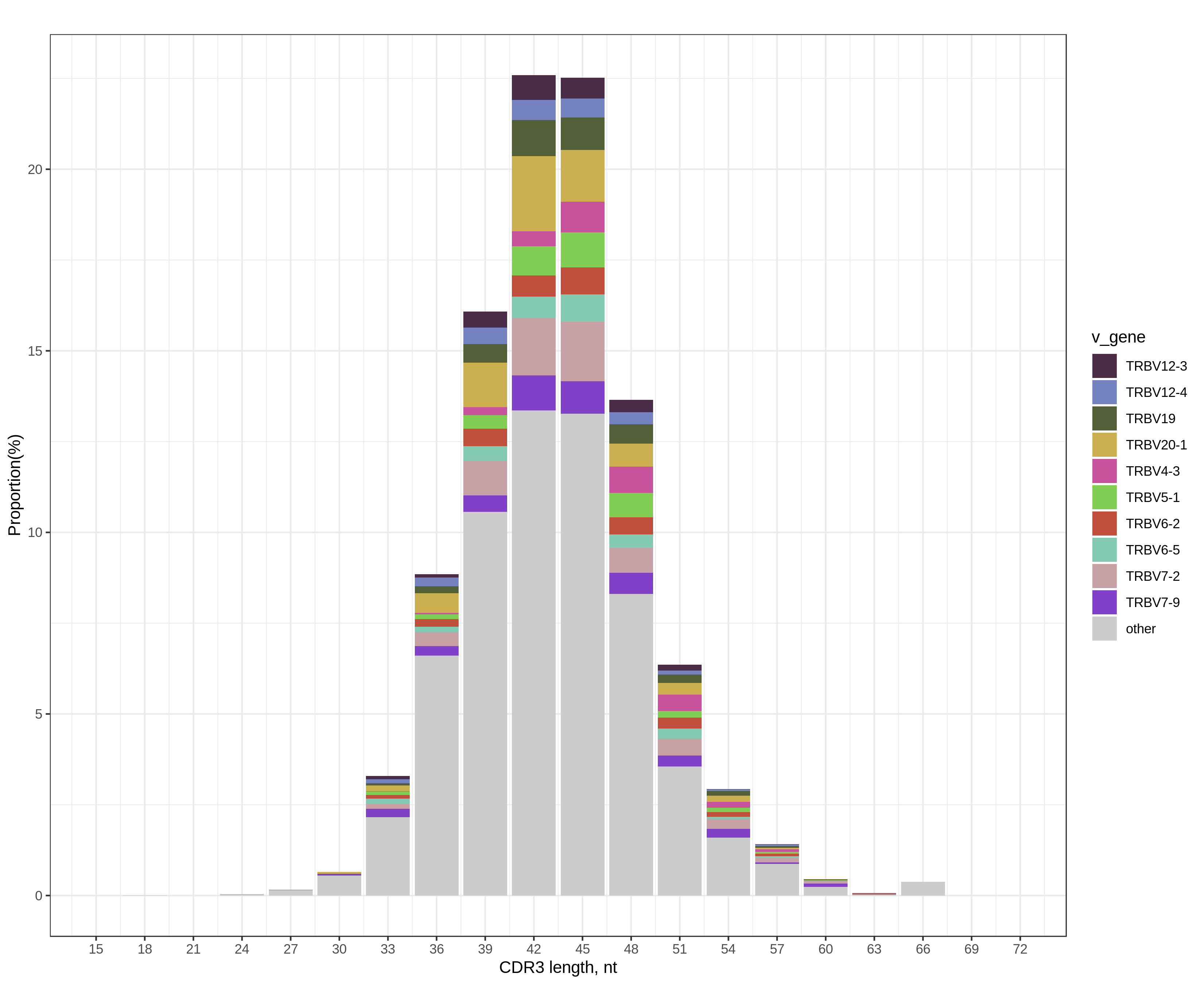
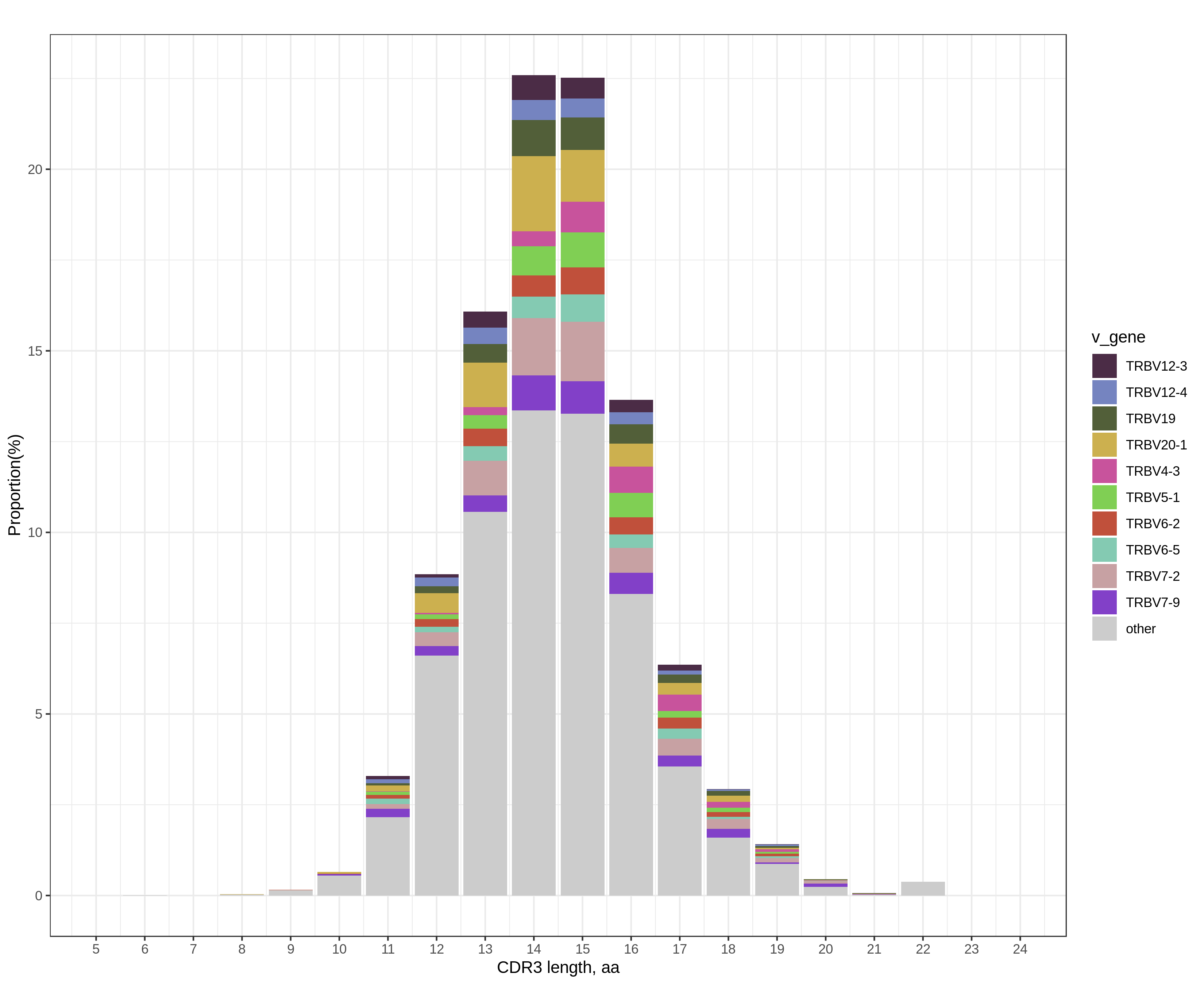
圖4. 組 Case VGene 核酸長度分布圖 圖5. 組 Case VGene 氨基酸長度分布圖
(左圖橫坐標為VGENE堿基序列長度,右圖橫坐標為VGENE氨基酸序列長度,縱坐標為clonotype的頻率,不同顏色表示不同clonotype,彩色表示top10 clonotype,灰色表示除top 10以外的其他clonotype。)
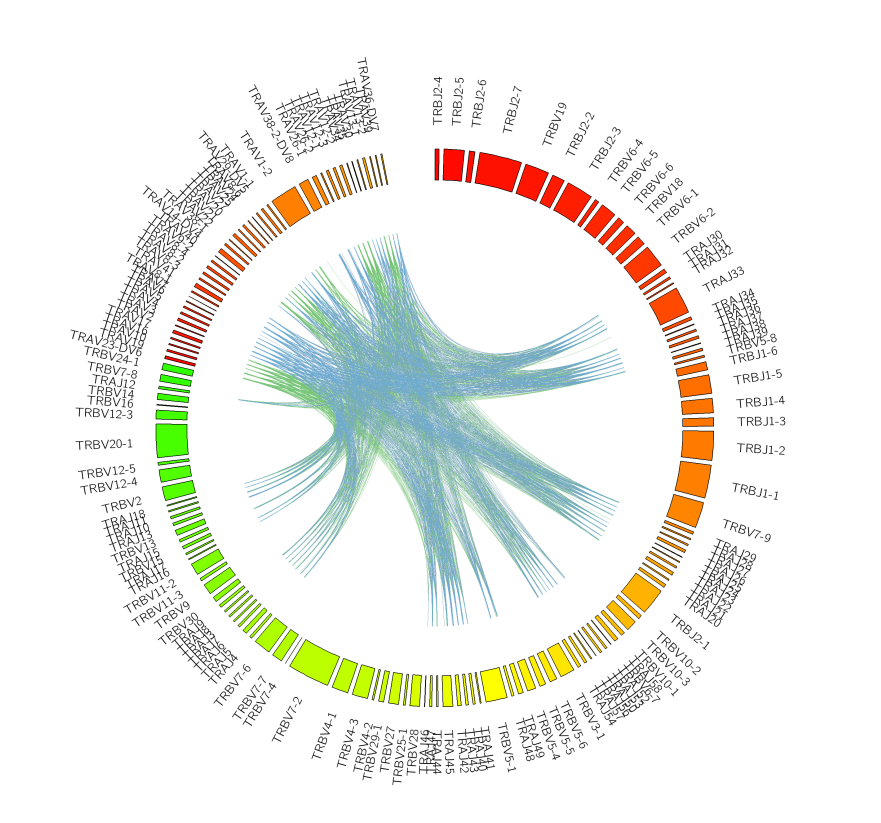
圖6. 樣品 TCR_Sample_1 鏈TRA_TRB V-J paired頻率Circos圖
(V-J paire頻率Circos圖,最外圈弧形表示V、J基因,每個顏色塊代表一種基因,基因頻率越高,色塊越寬;內部色塊間連接弧線表示V-J paired。)
5 技術優勢
可實現配對的重鏈和輕鏈(B細胞)或α和β鏈(T細胞)的全長測序;
精確到單細胞水平,能獲得大量單細胞的免疫組數據;
大規模單細胞VDJ表達譜測序,單個細胞成本大大降低;
同時獲得5’ gene expression的單細胞轉錄組數據,可與免疫組數據聯合分析
6 案例分享
Single-Cell Map of Diverse Immune Phenotypes in the Breast Tumor Microenvironment
發文期刊:Cell
影響因子: 30.41
了解腫瘤微環境中免疫細胞的表型,對于癌癥進展和免疫治療反應的研究至關重要。研究者使用單細胞RNA測序分析了來自8位原發性乳腺癌患者的45,000個免疫細胞,同時也添加了正常乳腺組織、血液和淋巴結中的免疫細胞作對照。研究者開發了預處理流程SeQC和貝葉斯聚類和歸一化方法Biscuit,用來解決單細胞數據固有的計算難題。研究中觀察到正常免疫細胞與腫瘤組織中免疫細胞之間有顯著相似性,但后者針對腫瘤微環境出現特異性連續表型擴展。對來自另外27,000個T細胞的成對單細胞RNA和T細胞受體(TCR)測序,結果揭示了TCR組合使用對表型多樣性的影響。研究結果支持T細胞連續活化模型,但不符合癌癥中的巨噬細胞極化模型[2]。本研究結果對表征腫瘤浸潤免疫細胞具有重要意義。
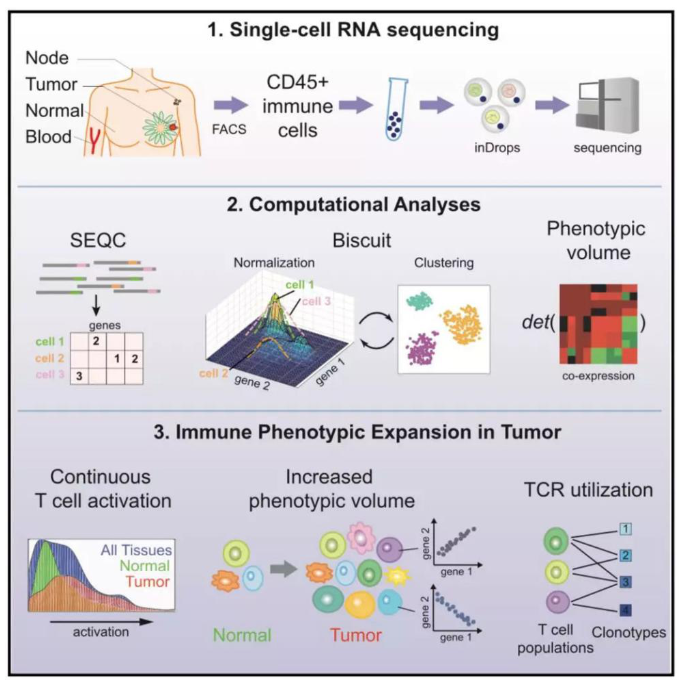
圖7 分析路程圖
參考文獻:
1. Yaari, G. and S.H. Kleinstein, Practical guidelines for B-cell receptor repertoire sequencing analysis. Genome medicine, 2015. 7(1): p. 1-14.
2. Azizi, E., et al., Single-cell map of diverse immune phenotypes in the breast tumor microenvironment. Cell, 2018. 174(5): p. 1293-1308. e36.