
傳統的高通量測序是對某一組織整體的細胞合集進行測序和分析,而某一個細胞的基因信息則會被整體覆蓋。2009年首個單細胞轉錄組測序技術問世,開啟了單細胞組學時代,在2013年單細胞測序技術被Nature Methods評為年度技術。單細胞測序技術經過十余年發展,各類單細胞測序技術平臺也如雨后春筍般出現,極大地推動生物醫學領域的相關研究,幫助科研人員克服了稀有生物樣本以及生物樣本內生異質性等重大挑戰。
DNBelab C4 基于液滴微流控原理,實現單細胞的捕獲和標記,使用DNBSEQ建庫技術構建專有文庫,采用DNBSEQ T7系統獲得測序數據,并使用配套的分析軟件進行數據分析與可視化,在單細胞層面進行基因表達、細胞注釋和異質化研究。
1、測序策略
測序策略:PE100
測序深度:推薦平均50k reads/cell
測序數據量:100~120G/樣本
2、送樣建議
(1)細胞:大于1*106個細胞;
(2)組織解離的細胞:大于1*106個細胞;
(3)血液:大于3 mL;
(4)PBMC:大于1*106個細胞;
(5)組織:大于黃豆粒大小;
(6)細胞活性大于80%。
3、技術優勢
(1)便攜,操作簡單:重量220g,無需電源,負壓驅動的液滴生成系統,有效地簡化液滴捕獲過程。
(2)雙磁珠高效捕獲:采用自主設計的捕獲磁珠,有效提高細胞捕獲效率以及獲取下撥的完整信息。
(3)低雙胞率,基因檢測能力更穩定:雙胞率低于8%,每個細胞平均基因數在1000~2000。
(4)數據高保真,性價比高:搭配國產超高通量DNBseq-T7測序平臺,既保證測序數據準確性,也降低測序成本。
4、數據分析
4.1 標準信息分析
(1)測序數據統計與評估
a. 各個樣本測序數據基本質控(reads數、測序飽和度等)
b. 各個樣本數據比對(細胞數目統計、reads基因組比對率等)
c. 多樣本數據合并(基因表達定量)
(2)單細胞亞群分類與分類結果可視化
a. 細胞過濾
b. 單細胞亞群分類(各亞群單個細胞基因中位數、各樣本在各亞群數目統計)
c. 分類結果可視化(聚類分析可視化(tSNE圖))
(3)亞群上調表達基因分析
a. 上調表達基因篩選
b. 上調表達基因基因GO功能富集分析
c. 上調表達基因KEGG功能富集分析
(4)標記基因篩選
a. 標記基因篩選(標記基因在各亞群平均表達量、標記基因聚類熱圖)
b. 各標記基因在群體中的表達分布(標記基因tSNE圖)
4.2 高級分析
(1)已知表達基因(分子markers或表面標記物)在各亞群表達分布圖及熱圖(甲方提供基因標準名稱)
(2)細胞軌跡分析
(3)加權基因共表達網絡(WGCNA)
(4)差異表達基因TF編碼能力預測
(5)差異表達基因蛋白互作分析
(6)基因相關性網絡分析
(7)細胞周期鑒定分析
5、技術流程
6、案例分析
案例(1) 非人靈長類動物食蟹猴的單細胞轉錄組圖譜
研究團隊對食蟹猴45個組織約114萬個細胞進行單細胞測序分析,獲得非人靈長類動物全身器官細胞圖譜(圖1)。基于該圖譜構建了126種病毒易感細胞類型的病毒數據庫,可以通過它快速查詢病毒最有可能侵染的細胞類型,同時看到該細胞類型可能分布的器官。該圖譜還可用于物種進化、人類疾病以及藥物評價和篩選相關的研究,為生物醫學的發展提供基礎性的資源和工具,為疾病診療、靶向藥物開發提供助力,為人類更好地探究生命的進化提供可能。
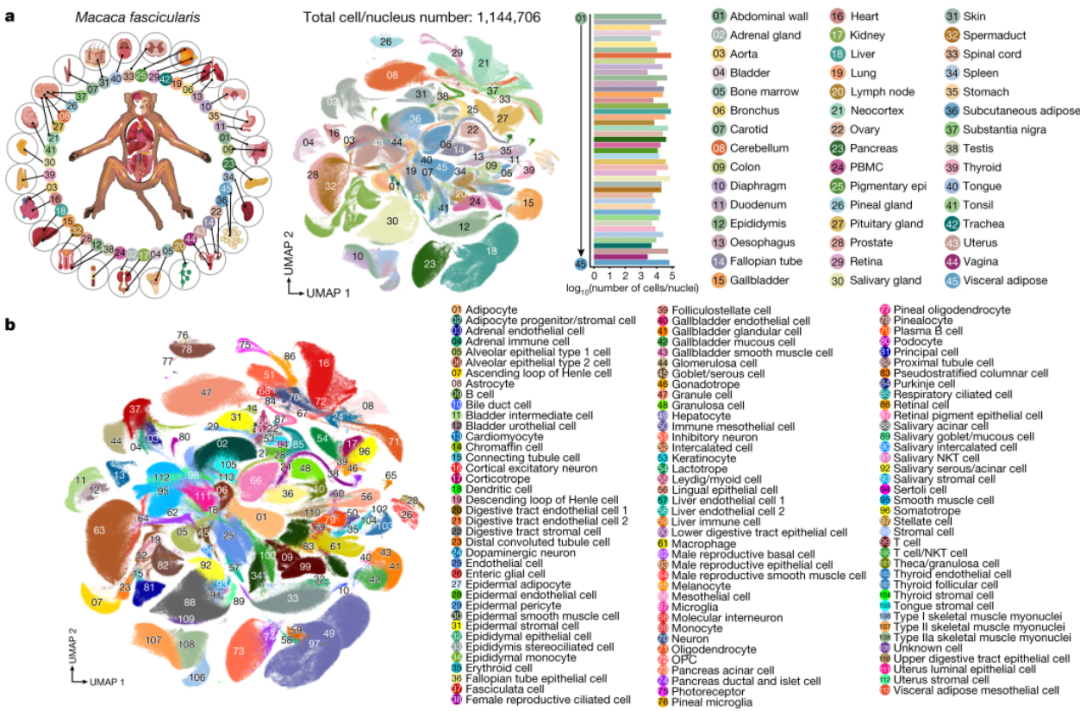
圖1 食蟹猴全身器官圖譜
案例(2) 人類多能性干細胞轉化為8細胞階段全能性胚胎樣細胞
研究團隊通過系統性的篩選,首次建立了體外全能性的8細胞期胚胎樣細胞(8CLC)的誘導和富集方法。基于DNBelab C4平臺,對8CLC誘導過程的細胞樣本進行了單細胞轉錄組(scRNA-seq)和染色質可及性(scATAC-seq)測序,詳細分析了8CLC群體的特征,并在多個誘導時間點描繪了8CLC群體的特征,并在多個誘導時間點描繪了8CLC誘導過程中轉錄組和染色質開放性的動態變化,為研究人類8細胞期的合子基因組激活和發育調控提供了重要的平臺和資源,將拓展我們對人類早期胚胎發育的有限認知。
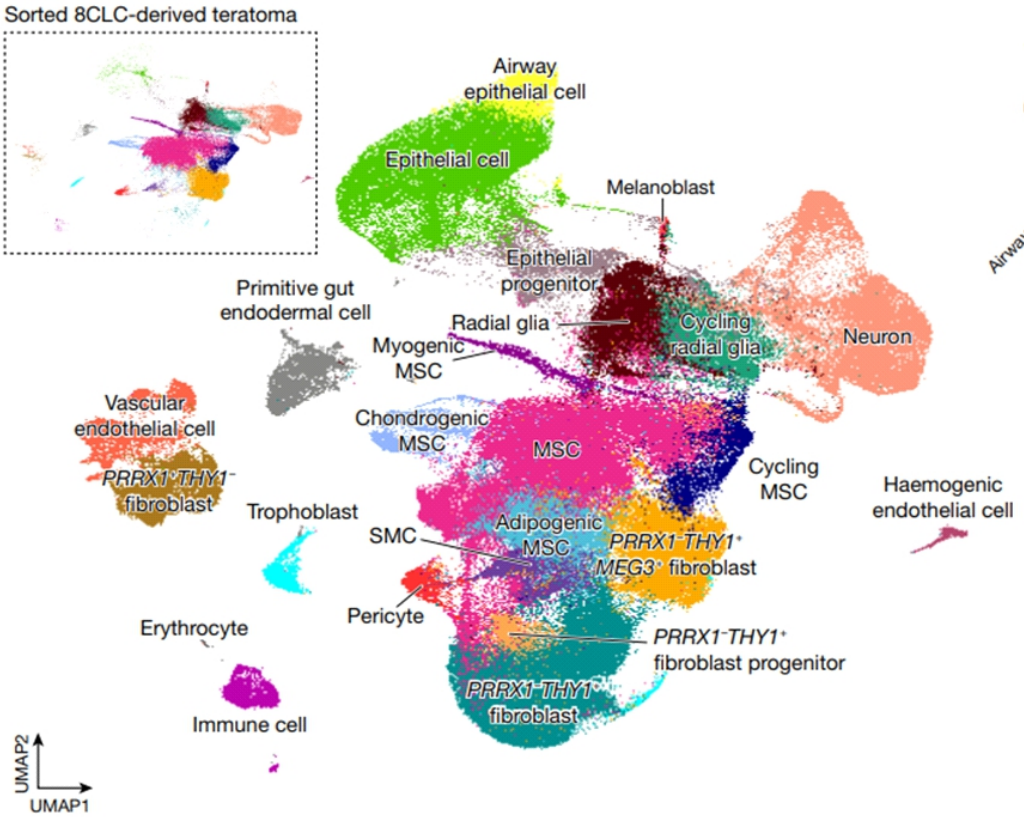
案例2 圖1 人胚胎干細胞圖譜
參考文獻:
[1]Han L,et al. Cell transcriptomic atlas of the non-human primateMacaca fascicularis[J]. Nature,2022
[2]Mazid, M.A., et al.Rolling back of human pluripotent stem cells to an 8-cell embryo-like stage[J]. Nature,2022